Implicaciones pronósticas de los polimorfismos relacionados con las dianas de los microRNAs en el Cáncer Colorrectal avanzado
José Carlos Villa Guzmán Servicio de Oncología Directores: Enrique Aranda Aguilar y Jesús García Foncillas. Universidad de Córdoba
RESUMEN
El cáncer colorrectal (CCR) es la tercera causa más frecuente de mortalidad por cáncer en los países occidentales. Los microRNAs (miRNAs) son pequeñas cadenas de RNA y controlan la traducción del RNA mensajero (mRNA) al unirse a su región 3’ UTR teniendo un importante papel en la regulación génica. Los miRNAs se han relacionado con el CCR estando alterada su regulación y comportándose como oncogenes o genes supresores de tumores. Los polimorfismos tipo SNP en la secuencia de la región 3’ UTR del mRNA relacionado con miRNAs contribuyen al desarrollo CCR.
La relevancia de estos polimorfismos en el CCR avanzado es poco conocida y en nuestro trabajo pretendemos estudiar las consecuencias pronósticas y predictivas de respuesta y de toxicidad al tratamiento antineoplásico.
Se han analizado 9 polimorfismos tipo SNP localizados en la región 3’ UTR de los genes APC, MTHFR, MTR, FGF2, TGBFR1, MMP2, CASP 7, GFI 1B y CYP 2C9 en 139 pacientes diagnosticados de cáncer colorrectal avanzado. Todos los pacientes han recibido una primera línea de tratamiento basada en fluoropirimidinas más oxaliplatino o irinotecán.
En los resultados hemos obtenido implicaciones de los polimorfismos en la supervivencia de los pacientes y en la respuesta y toxicidad desarrollada al tratamiento antineoplásico.
Los SNPs localizados en las regiones 3’ UTR del mRNA de genes relacionados con la etiopatogenia del CCR en nuestra serie se comportan como biomarcadores confiriendo un papel pronóstico o predictivo de respuesta y toxicidad al tratamiento antineoplásico.
INTRODUCCIÓN
El cáncer colorrectal (CCR) es la tercera neoplasia maligna en frecuencia y la segunda causa de muerte por cáncer en los países occidentales. El CCR es la segunda causa de muerte en los países desarrollados (figura 1). En el año 2009, se diagnosticaron 11233.700 nuevos casos de CCR a nivel mundial. Durante ese año, murieron 605.700 pacientes a causa del CCR en todo el mundo. Los pacientes con metástasis a distancia, es decir, con estadio IV, constituyen el 20-25% (figura 1).
Figura 1. 1A: Mortalidad asociada a cáncer a nivel mundial; 1B: Distribución de los estadios del CCR al diagnóstico.
En lo referente al tratamiento de esta enfermedad, la aparición del 5-FU, la modulación bioquímica, la infusión contínua de 5-FU, los esquemas de poliquimioterapia y actualmente los fármacos dirigidos contra dianas moleculares específicas han conseguido modificar la historia natural de esta neoplasia alcanzando medianas de supervivencia de hasta 30 meses según las series estudiadas. La tendencia actual en el manejo de esta entidad es personalizar el empleo de fármacos ofreciendo a los pacientes el tratamiento más activo y menos tóxico, de ahí la necesidad de tener biomarcadores fiables y reproducibles que nos permitan afianzar esta decisión. El ejemplo más representativo lo constituyen las mutaciones en el gen KRAS1, predictivas de respuesta a la terapia antiEGFR (cetuximab y panitumumab). Sin embargo, necesitamos nuevos biomarcadores que nos ayuden a seleccionar más a los pacientes para poder ofrecer el tratamiento menos tóxico y más activo.
En los tumores humanos existen numerosas alteraciones genéticas que se traducen en la pérdida de expresión de un gen supresor de tumores, la sobreexpresión de un oncogén o las alteraciones en la función de la proteína codificada. Estos hechos se pueden explicar por distintos mecanismos, entre los que se encuentran los polimorfismos genéticos y los microRNAs. Los miRNAs son moléculas de RNA, constituidas por una secuencia de 19 a 25 nucleótidos, capaces de regular la expresión génica de cientos de genes tanto a nivel de la transcripción como de la traducción2. Esta regulación la ejercen uniéndose a una secuencia específica del mRNA localizada en la región 3’ UTR, de tal manera que cuando esta unión es totalmente complementaria se acaba por destruir el mRNA y cuando no es totalmente complementaria se procede al silenciamiento del mRNA. Los miRNAs se han relacionado con la etiología, progresión y el pronóstico de distintos tumores. Además, dependiendo de los genes sobre los que actúan, los miRNAs pueden tener una función de oncogenes o genes supresores de tumores3.
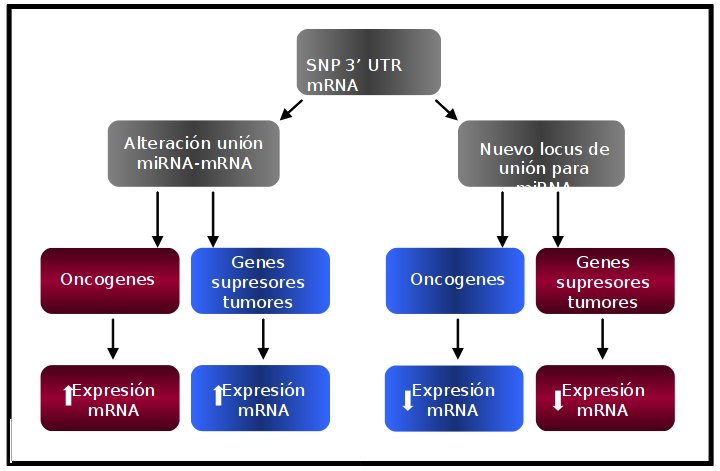
La alteración en la expresión de los miRNAs en los tumores y puede ser explicada por distintos hechos. Entre ellos figuran los polimorfismos tipo SNP localizados en los genes relacionados con los miRNAs4,5 de tal menara que pueden alterar la expresión del miRNA artefactando la unión miRNA-mRNA. Los SNPs localizados en la región 3’ UTR del mRNA pueden tener como consecuencia la destrucción del lugar de unión con el miRNA o crear nuevos “loci” sobre los que pueden interaccionar otros miRNAs alterando así la subsecuente traducción del mRNA6. Por tanto, los miRNAs juegan un papel primordial en la regulación del mRNA y los polimorfismos en la región 3’ UTR tienen consecuencias sobre esta regulación puesto que implican una alteración en la unión del miRNA-mRNA (aumentando la traducción del mRNA) o bien, la creación de un lugar nuevo de unión con el miRNA (disminuyendo la traducción de mRNA) (figura 2).
Figura 2: Consecuencias de los SNPs en la región 3’ UTR a nivel de la expresión del mismos según ocurra en oncogenes o genes supresores de tumores.
En lo referente al CCR, el papel de los SNPs relacionados con los microRNAs es poco conocido y existen escasos trabajos que diluciden la asociación entre los SNPs con la evolución del CCR7 y con la respuesta al tratamiento antineoplásico. Así pues, nuestro trabajo pretende identificar matices de esta relación en cuanto a factor pronóstico y predictivo.
HIPÓTESIS Y OBJETIVOS
Hipótesis:
Los polimorfismos tipo SNP localizados en la región 3’ UTR del RNA mensajero diana, lugar de unión de los microRNAs, podrían ser nuevos factores pronóstico y predictivos de respuesta al tratamiento antineoplásico en pacientes diagnosticados de carcinoma colorrectal avanzado.
Objetivos:
Los objetivos planteados en este trabajo son los siguientes:
-
Objetivo principal:
Establecer una correlación entre los polimorfismos localizados en la región 3’ UTR de los genes APC, CASP 7, FGF2, MMP2, MTHFR, MTR, TGFβR1, GFI 1B y CYP 2C9 con el tiempo a la progresión a la primera línea de tratamiento en pacientes diagnosticados de carcinoma colorrectal avanzado tratados con quimioterapia.
-
Objetivos secundarios:
Establecer correlación entre los polimorfismos estudiados y:
1. La respuesta al tratamiento antitumoral.
2. La duración de la respuesta obtenida a la quimioterapia.
3. La toxicidad desarrollada al tratamiento antineoplásico.
4. El intervalo libre de enfermedad en aquellos pacientes intervenidos.
5. La supervivencia global.
MATERIAL Y MÉTODOS
Para la realización de este estudio se recogieron 10 ml de sangre periférica de pacientes diagnosticados de carcinoma colorrectal metastático. El reclutamiento de los pacientes se realizó entre Mayo y Diciembre de 2008, teniendo en cuenta los siguientes criterios de inclusión:
- Diagnóstico histológico de carcinoma colorrectal;
- Estadio IV a la entrada del estudio;
- Haber recibido quimioterapia;
- Criterios homogéneos de seguimiento y valoración de respuesta según criterios de RECIST8 (Response Evaluation Criteria In Solid Tumors).
- Firmar el consentimiento informado para la inclusión en el estudio.
Por otro lado, los polimorfismos analizados en nuestro estudio tienen 2 características importantes:
-
Residir en la región 3’ UTR del mRNA.
-
Tener una frecuencia alélica menor reportada mayor del 20%.
El genotipado se realizará utilizando el método de discriminación alélicas y el de secuenciación. Los genes y los polimorfismos tipo SNP estudiados por discriminación alélica junto con las referencias de los assays (TaqMan®SNP Genotyping Assays) de la casa comercial Applied Biosystem, el cambio de nucleótido y la función del polimorfismo que se van a analizar están reportados en la tabla 1.
|
NOMBRE GEN |
SNP |
POSICIÓN |
CAMBIO NUCLEOTIDO |
mirRNA asociado |
REF Applied Biosystem |
|
APC |
3´UTR |
A>G |
miR-642 |
C_1987140_10 |
|
|
MTHFR
|
3´UTR |
T>G |
miR-555 |
C_27968106_10 |
|
|
MTR
|
3´UTR |
A>C |
miR-380-5p, miR-563 |
C_26588244_10 |
|
|
FGF2 |
3´UTR |
T>C |
miR-545 |
C_8837778_10 |
|
|
TGFBR1 |
3´UTR |
C>G |
miR-511 |
C_662618_1 |
|
|
MMP2
|
3´UTR |
A>C |
miR-520 |
C_3225976_10 |
|
|
CASP7
|
3´UTR |
T>C |
miR-224 |
C_500777_10 |
Tabla 1: Características de los SNPs analizados por discriminación alélica.
Los genes y polimorfismos que serán analizados por secuenciación junto con las características antes enumeradas las podemos observar en la tabla 2.
|
NOMBRE GEN |
SNP |
POSICION |
miRNA asociado |
CAMBIO NUCLEOTIDO |
|
GFI 1B |
3´UTR |
miR-484 |
G>C |
|
|
CYP 2C9 |
3´UTR |
miR-521 |
G>A |
Tabla 2: Características de los SNPs analizados por secuenciación.
RESULTADOS
1.- Asociación de los polimorfismos con las respuestas y la duración de respuesta a la quimioterapia.
En la tabla 3 podemos observar la asociación de los distintos polimorfismos con las respuestas a la quimioterapia en general. El gen APC mantiene una relación con las respuestas objetivas y la tasa de control de enfermedad (p=0,006 y 0,02 respectivamente).
|
RESPUESTAS OBJETIVAS |
TASA DE CONTROL DE ENFERMEDAD |
||||
|
Genes |
SNP |
n (%) |
p |
n (%) |
p |
|
APC |
GG GA AA |
12 (85,7) 28 (46,7) 26 (45,6) |
0,006 |
14 (100) 56 (93,3) 45 (78,9) |
0,02 |
|
MTHFR |
GG GT TT |
29 (46,0) 29 (54,7) 8 (53,3) |
0,81 |
55 (87,3) 46 (86,8) 14 (93,3) |
0,78 |
|
MTR |
CC CA AA |
23 (48,9) 31 (51,7) 12 (50,0) |
0,96 |
41 (87,2) 52 (86,7) 22 (91,7) |
0,81 |
|
FGF2 |
CC CT TT |
42 (46,2) 23 (60,5) 1 (50,0) |
0,53 |
80 (87,9) 33 (86,8) 2 (100) |
0,85 |
|
TGFβR1 |
GG GC CC |
39 (51,3) 26 (54,2) 1 (14,3) |
0,23 |
69 (90,8) 41 (85,4) 5 (71,4) |
0,26 |
|
MMP2 |
CC CA AA |
12 (50,0) 24 (47,3) 30 (60,2) |
0,051 |
20 (83,3) 54 (91,5) 41 (85,4) |
0,48 |
|
CASP7 |
CC CT TT |
4 (66,7) 24 (43,6) 38 (54,3) |
0,44 |
6 (100) 49 (89,1) 60 (85,7) |
0,54 |
|
GFI 1B |
CC CG GG |
7 (47,7) 34 (63,0) 25 (40,0) |
0,11 |
13 (86,7) 51 (94,4) 51 (82,3) |
0,13 |
|
CYP 2C9 |
AA AG GG |
38 (50,0) 23 (50,0) 5 (55,6) |
0,83 |
67 (87,2) 41 (89,1) 7 (77,8) |
0,62 |
Tabla 3: Asociación de los polimorfismos estudiados y la respuesta a la quimioterapia mediante las pruebas de χ2. Respuestas objetivas: suma de las respuestas completas (RC) más las respuestas parciales (RP). Tasa de control de enfermedad: suma de RC más RP más estabilización de enfermedad (EE).
A continuación, detallamos la relación de los distintos polimorfismos con la respuesta obtenida a la quimioterapia en función del régimen empleado. De nuevo, el gen APC está implicado con las respuestas a la quimioterapia, siendo significativo en los pacientes tratados con regímenes basados en irinotecán (tabla 4).
|
Población total (n=139) |
Reg. Oxaliplatino (n=67) |
Reg. Irinotecán (n=58) |
Fluoropirimidinas (n=14) |
|
|
Genes |
RO (p) TCE (p) |
RO (p) TCE (p) |
RO (p) TCE (p) |
RO (p) TCE (p) |
|
APC |
0,006 0,02 |
0,139 0,66 |
0,098 0,021 |
0,63 0,47 |
|
MTHFR |
0,81 0,78 |
0,68 0,76 |
0,73 0,84 |
0,54 0,72 |
|
MTR |
0,96 0,81 |
0,93 0,91 |
0,77 0,41 |
0,87 0,70 |
|
FGF2 |
0,53 0,85 |
0,44 0,92 |
0,65 0,77 |
0,27 0,10 |
|
TGFβR1 |
0,23 0,26 |
0,75 0,50 |
0,22 0,10 |
0,16 0,72 |
|
MMP2 |
0,051 0,48 |
0,42 0,71 |
0,28 0,94 |
0,19 0,18 |
|
CASP7 |
0,44 0,54 |
0,50 0,76 |
0,95 0,79 |
0,21 0,15 |
|
GFI 1B |
0,11 0,13 |
0,34 0,55 |
0,19 0,38 |
0,21 0,07 |
|
CYP 2C9 |
0,83 0,62 |
0,15 0,78 |
0,27 0,45 |
0,21 0,42 |
Tabla 4: Asociación de los polimorfismos estudiados y la respuesta a la quimioterapia en función del régimen de tratamiento empleado mediante las pruebas de χ2. RO: respuestas objetivas o suma de las respuestas completas (RC) más las respuestas parciales (RP). TCO: tasa de control de enfermedad o suma de RC más RP más estabilización de enfermedad (EE).
Finalmente, resumimos las distintas medianas de duración de respuesta a la quimioterapia según el polimorfismo de cada gen y los regímenes de quimioterapia empleados. El gen CYP 2C9 parece estar implicado en la duración de la respuesta obtenida con la quimioterapia independientemente del esquema utilizado al igual que el gen MPP2 (tabla 5).
|
Población total (n=139)
|
Reg. Oxaliplatino (n=67) |
Reg. Irinotecán (n=58) |
Fluoropirimidinas (n=14) |
||
|
Genes
|
SNP |
Meses, IC95% p | Meses, IC95% p | Meses, IC95% p | Meses, IC95% p |
|
APC |
GG GA AA |
5 [2,85 – 7,14] 6 [2,65 – 9,34] 0,46 3 [0,00 – 6,09] |
5 [0,10 – 9,90] 6 ** 0,17 3 [1,71 – 4,28] |
* 3 [0,00 – 6,20] 0,64 12 ** |
4 ** 2 ** 0,80 3 ** |
|
MTHFR |
GG GT TT |
5 [0,00 – 10,84] 6 [0,50 – 11,50] 0,37 9 [0,00 – 20,76] |
5 [1,08 – 8,92] 6 [0,45 – 11,54] 0,25 9 [0,00 – 18,60] |
3 ** 12 [0,00 – 39,94] 0,64 *** |
2 ** 3 **0,031 16 ** |
|
MTR |
CC CA AA |
3 [2,24 – 3,75] 7 [3,33 – 10,76] 0,92 3 [1,40 – 4,60] |
3 [1,86 – 4,13] 8 [5,07 – 10,92] 0,009 *** |
3 ** 12 [2,39 – 21,60] 0,77 2 ** |
16** 2** 0,40 3** |
|
FGF2 |
CC CT TT |
6 [4,62 – 7,37] 3 [1,53 – 4,46] 0,48 * |
6 [3,84 – 8,15] 3 [0,00 – 7,90] 0,92 * |
6 [0,00 – 32,46] 3 [0,00 – 12,80] 0,22 *** |
4 [2,40 – 5,60] 2** 0,083 *** |
|
TGFβR1 |
GG GC CC |
6 [0,55 –14,44] 6 [2,47 – 5,52] 0,63 * |
3 [1,71 – 4,28] 7 [4,22 – 9,77] 0,084 * |
12 [0,00 – 24,88] 3 ** 0,85 *** |
3** 2** 0,52 *** |
|
MMP2 |
CC CA AA |
9 [0,00 – 21,88] 3 [1,65 – 4,34] 0,004 7 [4,55 – 9,44] |
9 [0,00 – 18,60] 3 [0,85 – 5,14] 0,011 7 [5,71 – 8,28] |
3 ** 6 ** 0,55 24 [0,00 – 69,08] |
* 2** 0,09 4** |
|
CASP7 |
CC CT TT |
5 ** 6 [0,15 – 11,84] 0,68 6 [1,96 – 10,03] |
5 ** 6 [0,12 – 11,88] 0,64 6 [1,86 – 10,13] |
*** 3 [1,40 – 4,60] 0,84 12 [0,00 – 24,84] |
*** 4** 0,13 2** |
|
GFI 1B |
CC CG GG |
3 [0,00 – 7,90] 3 [0,73 – 5,26] 0,43 7 [3,76 – 10,23] |
3 ** 6 [0,00 – 12,44] 0,79 6 [3,22 – 12,44] |
2 ** 3 ** 0,18 30 ** |
*** 3 [1,40 – 4,60] 0,91 4 ** |
|
CYP 2C9 |
AA AG GG |
6 [3,10 – 8,89] 15 [9,45– 20,54] 0,002 2 ** |
6 [4,30 – 7,69] 3 ** 0,029 2 ** |
3 [2,15 – 3,84] 24 [6,36 – 41,64] 0,007 *** |
2 ** 3 ** 0,21 4 ** |
Tabla 5: Asociación de los polimorfismos con la mediana de duración de respuesta a la quimioterapia en función del esquema empleado según el método de Kaplan-Meier.* Estadísticos no calculados debido al que sólo hay un paciente incluido en ese grupo. ** Límites no calculados debido al escaso número de pacientes.***No existen pacientes en este grupo.
2.- Asociación de los polimorfismos con la toxicidad desarrollada con la quimioterapia.
En este apartado resumiremos las distintas toxicidades aparecidas con la quimioterapia y los distintos polimorfismos genéticos. Con la diarrea se asocian los genes MTHFR y MTR con significación estadística (p= 0,029 y 0,037 respectivamente). Por otro lado, el gen FGF 2 está implicado en el desarrollo de toxicidad hematológica y a nivel de mucosas en nuestra población con un nivel de significación de 0,0001 y 0,005 respectivamente. Por último, también hemos encontrado asociación entre el gen CASP7 y la neurotoxicidad (p=0,0001) (tabla 6).
|
Hematológica |
Digestiva |
Mucosas |
Neurológica |
Total (p) |
||||
|
Genes |
Neutropenia (p) |
Anemia (p) |
Naúseas (p) |
Vómitos (p) |
Diarrea (p) |
Mucositis (p) |
Parestesias (p) |
|
|
APC |
0,32 |
0,55 |
0,30 |
0,46 |
0,33 |
0,48 |
0,094 |
0,49 |
|
MTHFR |
0,16 |
0,59 |
0,34 |
0,74 |
0,029 |
0,22 |
0,55 |
0,52 |
|
MTR |
0,28 |
0,41 |
0,23 |
0,33 |
0,037 |
0,11 |
0,45 |
0,31 |
|
FGF2 |
0,003 |
0,0001 |
0,77 |
0,60 |
0,89 |
0,005 |
0,37 |
0,13 |
|
TGFβR1 |
0,18 |
0,70 |
0,88 |
0,76 |
0,23 |
0,82 |
0,24 |
0,34 |
|
MMP2 |
0,28 |
0,11 |
0,38 |
0,72 |
0,60 |
0,93 |
0,64 |
0,49 |
|
CASP7 |
0,52 |
0,50 |
0,25 |
0,40 |
0,63 |
0,44 |
0,0001 |
0,69 |
|
GFI 1B |
0,49 |
0,54 |
0,87 |
0,45 |
0,32 |
0,66 |
0,41 |
0,044 |
|
CYP2C9 |
0,20 |
0,69 |
0,47 |
0,72 |
0,46 |
0,47 |
0,73 |
0,94 |
Tabla 6 : Asociación de los polimorfismos genéticos con las distintas toxicidades en la población total (n=139).
A continuación, en la siguiente tabla resumimos las distintas toxicidades en función del régimen de quimioterapia empleado (tabla 7).
|
Tabla 8.A |
Tipo de toxicidad grado III/IV en reg. oxaliplatino (n=67) |
|||||||
|
Hematológica |
Digestiva |
Mucosas |
Neurológica |
Total (p) |
||||
|
Neutropenia (p) |
Anemia (p) |
Naúseas (p) |
Vómitos (p) |
Diarrea (p) |
Mucositis (p) |
Parestesias (p) |
||
|
APC |
0,48 |
0,51 |
0,51 |
0,44 |
0,26 |
0,61 |
0,24 |
034 |
|
MTHFR |
0,78 |
0,53 |
0,53 |
0,53 |
0,13 |
0,19 |
0,58 |
0,76 |
|
MTR |
0,43 |
0,37 |
0,37 |
0,098 |
0,077 |
0,42 |
0,50 |
0,58 |
|
FGF2 |
0,0001 |
0,0001 |
0,81 |
0,81 |
0,33 |
0,005 |
0,33 |
0,26 |
|
TGFβR1 |
0,18 |
0,70 |
0,88 |
0,76 |
0,23 |
0,82 |
0,24 |
0,34 |
|
MMP2 |
0,26 |
0,098 |
0,55 |
0,41 |
0,48 |
0,77 |
0,58 |
0,49 |
|
CASP7 |
0,50 |
0,37 |
0,37 |
0,71 |
0,79 |
0,47 |
0,003 |
0,40 |
|
GFI 1B |
0,70 |
0,59 |
0,59 |
0,59 |
0,57 |
0,63 |
0,33 |
0,25 |
|
CYP2C9 |
0,64 |
0,72 |
0,72 |
0,72 |
0,50 |
0,20 |
0,71 |
0,31 |
|
Tabla 8.B |
Tipo de toxicidad grado III/IV en reg. irinotecan (n=58) |
|||||||
|
Hematológica |
Digestiva |
Mucosas |
Neurológica |
Total |
||||
|
Neutropenia (p) |
Anemia (p) |
Naúseas (p) |
Vómitos (p) |
Diarrea (p) |
Mucositis (p) |
Parestesias (p) |
(p) |
|
|
APC |
0,35 |
– |
0,62 |
0,22 |
0,40 |
0,81 |
– |
0,55 |
|
MTHFR |
0,019 |
– |
0,69 |
0,83 |
0,30 |
0,26 |
– |
0,51 |
|
MTR |
0,68 |
– |
0,042 |
0,58 |
0,68 |
0,18 |
– |
0,63 |
|
FGF2 |
0,67 |
– |
0,16 |
0,22 |
0,47 |
0,22 |
– |
0,41 |
|
TGFβR1 |
0,19 |
– |
0,76 |
0,43 |
0,21 |
0,43 |
– |
0,29 |
|
MMP2 |
0,071 |
– |
0,11 |
0,80 |
0,71 |
0,62 |
– |
0,17 |
|
CASP7 |
0,48 |
– |
0,60 |
0,20 |
0,84 |
0,81 |
– |
0,74 |
|
GFI 1B |
0,70 |
– |
0,48 |
0,66 |
0,23 |
0,66 |
– |
0,15 |
|
CYP2C9 |
0,022 |
– |
0,69 |
0,92 |
0,096 |
0,36 |
– |
0,010 |
| Tabla 9.C |
Tipo de toxicidad grado III/IV en reg. fluoropirimidinas (n=14) |
|||||||
|
Hematológica |
Digestiva |
Mucosas |
Neurológica |
Total (p) |
||||
|
Neutropenia (p) |
Anemia (p) |
Naúseas (p) |
Vómitos (p) |
Diarrea (p) |
Mucositis (p) |
Parestesias (p) |
||
|
APC |
– |
– |
– |
– |
0,84 |
– |
– |
0,84 |
|
MTHFR |
– |
– |
– |
– |
0,58 |
– |
– |
0,58 |
|
MTR |
– |
– |
– |
– |
0,38 |
– |
– |
0,38 |
|
FGF2 |
– |
– |
– |
– |
0,42 |
– |
– |
0,42 |
|
TGFβR1 |
– |
– |
– |
– |
0,40 |
– |
– |
0,40 |
|
MMP2 |
– |
– |
– |
– |
0,28 |
– |
– |
0,28 |
|
CASP7 |
– |
– |
– |
– |
0,51 |
– |
– |
0,51 |
|
GFI 1B |
– |
– |
– |
– |
0,23 |
– |
– |
0,23 |
|
CYP2C9 |
– |
– |
– |
– |
0,51 |
– |
– |
0,51 |
Tablas 9: Asociación de los distintos polimorfismos genéticos en función del esquema de quimioterapia utilizado. 9.A regímenes basados en oxaliplatino, 9.B regímenes basados en irinotecán y 9.C regímenes basados en fluoropirimidinas en monoterapia.
En aquellos pacientes que reciben quimioterapia basada en oxaliplatino, de nuevo el gen FGF2 está implicado en el desarrollo de toxicidad hematológica (p=0,0001) y a nivel de mucosas (p=0,005) y el gen CASP7 interviene en la aparición de neurotoxicidad (p=0,003) en los pacientes de nuestra serie tratados con oxaliplatino.
En cambio, en los pacientes que reciben esquemas basados en irinotecán, los genes MTHFR y CYP 2C9 participan en la aparición de toxicidad hematológica con significación estadística, p=0,019 y 0,022 respectivamente.
Finalmente, no hemos encontrado asociaciones entre los polimorfismos genéticos estudiados y la aparición de toxicidad cuando se emplean fluoropirimidinas en monoterapia.
3.- Asociación de los polimorfismos con los intervalos libres de enfermedad.
En este apartado resumimos las distintas medianas del intervalo libre de enfermedad en pacientes intervenidos de CCR estadio II/III. Las variantes genéticas homocigóticas mutadas de los genes APC y TGFβR1 presentan unas medianas menores existiendo significación estadística (p=0,018 y 0,0001 respectivamente).
|
Intervalo libre de enfermedad |
|||
|
Genes |
SNP |
Meses, [IC95%] |
p |
|
APC |
GG GA AA |
15 [9,86 – 20,13] 17 [10,35 – 23,64] 13 [8,30 – 17,69] |
0,023 |
|
MTHFR |
GG GT TT |
14 [7,29 – 20,71] 12 [7,78 – 16,21] 20 [14,86– 25,13] |
0,19 |
|
MTR |
CC CA AA |
12 [7,86 – 16,13] 16 [13,47 – 18,53] 20 [8,67 – 31,32] |
0,28 |
|
FGF2 |
CC CT TT |
16 [12,26 – 19,73] 12 [9,16 – 14,64] * |
0,47 |
|
TGFβR1 |
GG GC CC |
13 [9,30 – 16,69] 16 [10,66 – 21,33] 7 [4,85 – 9,14] |
0,006 |
|
MMP2 |
CC CA AA |
20 [4,72 – 35,27] 13 [10,08 – 15,91] 12 [3,46 – 20,53] |
0,09 |
|
CASP7 |
CC CT TT |
17 [10,55 – 23,44] 15 [10,0 – 19,99] 12 [9,22 – 14,78] |
0,95 |
|
GFI 1B |
CC CG GG |
12 [7,70 – 16,29] 15 [9,91 – 20,80] 13 [8,19 – 17,80] |
0,89 |
|
CYP 2C9 |
AA AG GG |
12 [8,30 – 15,69] 15 [9,15 – 20,84] 16 [5,54 – 26,45] |
0,74 |
Tabla 10: Asociación de los distintos polimorfismos con el intervalo libre de enfermedad. *Estadísticos no calculados por no existir pacientes en este grupo.
4.- Asociación de los polimorfismos con el intervalo libre de progresión.
A continuación resumimos las distintas medianas de supervivencia libre de progresión para las distintas variantes de los genes estudiados y según el régimen de quimioterapia empleado. Cuando analizamos la población general, las variantes homocigóticas mutadas de los genes TGFβR1 y de CYP 2C9 presentan un menor intervalo libre de progresión con significación estadística (p=0,008 y 0,014 respectivamente). Este hecho se repite en los pacientes que reciben oxaliplatino, irinotecán o fluoropirimidinas, no obstante la significación estadística se alcanza en el grupo de pacientes que reciben un esquema de quimioterapia basado en irinotecán (tabla 11).
|
Población total (n=139) |
Reg. Oxaliplatino (n=67) |
Reg. Irinotecán (n=58) |
Fluoropirimidinas (n=14) |
||
|
Genes |
SNP | Meses, IC95% p | Meses, IC95% p | Meses, IC95% p | Meses, IC95% p |
|
APC |
GG GA AA |
12 ** 10 [8,26 – 11,73] 0,87 11 [7,55 – 14,44] |
12 ** 10 [7,57 – 12,52] 0,34 9 [3,58 – 14,41] |
* *0,79 * |
* 8 [4,70 – 11,20] 0,79 9 [0,00 – 18,96] |
|
MTHFR |
GG GT TT |
10 [8,40 – 11,59] 9 [5,94 – 12,05] 0,65 4 [4,00 – 27,99] |
10 [8,89 – 11,10] 12 [9,33 – 14,66] 0,67 6 [0,00 – 16,57] |
11 [8,17 – 13,82] 9 [0,00 – 18,79] 0,53 16 [0,00 – 36,73] |
8** 6 [3,82 – 8,17] 0,33 21** |
|
MTR |
CC CA AA |
9 [7,01 – 10,98] 12 [9,99 – 14,00] 0,74 9 [5,11 – 12,88] |
9 [4,80 – 13,19] 12 [10,43–13,56] 0,35 * |
8 [6,76 – 9,23] 16 [8,50 – 23,49] 0,45 17 [8,31 – 25,69] |
21 [0,00 – 47,51] 6 ** 0,78 9 [2,36 – 15,63] |
|
FGF2 |
CC CT TT |
10 [8,08 – 11,91] 11 [5,72 – 10,27] 0,74 4 ** |
10 [8,11 – 11,88] * 0,64 * |
12 [6,26 – 17,37] 11 [1,84 – 20,15] 0,72 * |
9 [4,70 – 13,29] 2 ** 0,20 * |
|
TGFβR1 |
GG GC CC |
12 [9,11 –14,88] 10 [7,88–12,11] 0,008 6[2,65 – 9,34] |
* * 0,82 * |
14 [6,21 – 21,78] 16 [5,43– 26,56] 0,046 3 [ 0,00 – 5,99] |
21 [2,46–29,53] 6** 0,21 4** |
|
MMP2 |
CC CA AA |
11 [3,54 – 18,45] 10 [8,03 – 11,96] 0,54 12 [6,18 – 17,81] |
6 [0,00 – 12,45] 10 [8,91 – 11,08] 0,77 12 [9,23 – 14,77] |
11 [5,24 – 16,75] 11 [5,51 – 16,48] 0,67 19 [2,17 – 35,82] |
1 ** 8 [3,82 –12,17] 0,050 6 [ 0,00 –18,41] |
|
CASP7 |
CC CT TT |
11 [0,52 – 21,47] 11 [8,77 – 13,22] 0,68 10 [7,54 – 12,45] |
11 ** 10 [4,64 – 15,35] 0,41 9 [6,63 – 11,37] |
4** 11 [8,19 – 13,80] 0,92 14 [7,99 – 20,00] |
* 6 [0,00 – 17,10] 0,63 8 [1,48 – 14,51] |
|
GFI 1B |
CC CG GG |
12 [7,13 – 16,86] 11 [7,05 – 14,94] 0,29 10 [5,80 – 14,19] |
10 [4,12 – 15,88] 10 [6,16 – 17,83] 0,27 10 [2,62 – 17,37] |
9 [0,39 – 17,60] 11 [7,97 – 14,02] 0,67 4 [1,83 – 22,16] |
* * 0,012 * |
|
CYP 2C9 |
AA AG GG |
9 [6,99 – 11,00] 16 [9,95–20,06] 0,014 6 [2,20 – 9,80] |
11 [7,55 – 14,45] 10 [8,03 – 19,97] 0,88 * |
9 [6,64 – 11,35] 19 [11,7–26,24] 0,074 * |
6 [0,01 – 12,98] 21 [2,28 – 39,71] 0,10 6 ** |
Tabla 11: Asociación de los distintos polimorfismos con el intervalo libre de progresión y los distintos regímenes de quimioterapia empleados. *Estadísticos no calculados por no existir pacientes en este grupo. **Límites no calculados debido a que sólo hay un paciente.
DISCUSIÓN:
Los microRNAs son pequeñas secuencias de RNA con una implicación importante en la etiopatogenia del CCR, consecuencia de una alteración en su expresión existe una alteración en la expresión de oncogenes y de genes supresores de tumores. Los polimorfismos tipo SNP localizados en las regiones diana 3’ UTR de los microRNAs justifican en parte la alteración en la función de los microRNAs en los tumores. Teniendo en cuenta el carácter retrospectivo de nuestro estudio, hemos observado que los polimorfismos localizados en la región 3’ UTR de genes implicados en la etiopatogenia del CCR se comportan como noveles biomarcadores, relacionándose con el tiempo a la progresión (objetivo primario) y con la respuesta, duración de la respuesta y toxicidad desarrollada al tratamiento citostático (objetivo secundario).
Si tenemos en cuenta la fisiología del microRNA cuando se une a la región 3’ UTR del mRNA controlando su expresión (degradándolo si la unión es perfecta o silenciando su traducción en el ribosoma si la unión es parcial), estos SNPs localizados en dicha región 3’UTR del mRNA pueden alterar la función reguladora del miRNA suponiendo una alteración en la regulación génica a nivel pos-trascripcional.
CONCLUSIONES:
Teniendo en cuenta nuestros resultados, podemos concluir que los SNPs localizados en las regiones 3’ UTR del mRNA, diana del miRNA, de genes relacionados con la etiopatogenia del CCR en nuestra serie:
- Se comportan como biomarcadores confiriendo un papel pronóstico o predictivo de respuesta y toxicidad al tratamiento antineoplásico.
-
Los SNPs de los genes TGFβR1, FGF2 y CYP 2C9 presentan un papel pronóstico explicando diferencias en el intervalo libre de enfermedad (APC y TGFβR1), en el intervalo libre de progresión (TGFβR1 y CYP 2C9).
-
El SNP del gen APC es factor predictivo de respuesta y los SNPs MMP2 y CPY2C9 de duración de respuesta a la quimioterapia.
-
Los SNPs de los genes MTHFR, MTR, FGF2 y CASP 7 son factores predictivos de toxicidad a la quimioterapia.
-
Son necesarios estudios prospectivos que confirmen los resultados obtenidos en este trabajo así como el papel de estos SNPs en cáncer de colon localmente avanzado y metastásico.
BIBLIOGRAFÍA
1.- Karapetis CS , S Khambata -Ford , DJ Jonker et al. K-ras mutations and benefit from cetuximab in advanced colorectal cancer. N Engl J Med 2008; 359 (17): 1757-1765
2.- Bartel DP. MicroRNAs: genomics, biogenesis, mechanism, and function. Cell 2004; 116 (2): 281-97.
3.- Carleton M, Cleary MA, Linsley PS. MicroRNAs and cell cycle regulation. Cell Cycle 2007; 6 (17): 2127-32.
4.- Chen K, Rajewsky N. Natural selection on human microRNA binding sites inferred from SNP data. Nat Genet 2006; 38 (12): 1452-6.
5.- Saunders MA, Liang H, and Li WH. Human polymorphism at microRNAs and microRNA target sites. Proc Natl Acad Sci U S A 2007; 104 (9): 3300-5.
6.- Yu Z, Li Z, Jolicoeur N, Zhang L et al. Aberrant allele frequencies of the SNPs located in microRNA target sites are potentially associated with human cancers. Nucleic Acids Res 2007; 35 (13): 4535-41.
7.- Landi D, Gemignani F, Naccarati A et al. Polymorphisms within micro-RNA-binding sites and risk of sporadic colorectal cancer. Carcinogenesis 2008; 29 (3): 579-84.
8.- Greene F, Page D, Fleming I et al. AJCC Cancer Staging Manual. New York: Springer-Verlag; 2002.















Palabras Clave: biomarcadores, carcinoma colorrectal, microRNAs, polimorfismos, regulación génica